転写が活性化される状態では、特徴的な高次ゲノム構造やタンパク質の凝集体が形成されること、さらに遺伝子の周辺粘度が上昇し、エンハンサーとプロモーターの相互作用時間が延長することが観察されました。この研究は、大石さん(当ラボ)、新海創也さん(理研)、大浪修一さん(理研)、細田一史さん(あんさんが研究舎)、大川恭行さん(九大生医研)との共同研究によるものです!
https://www.biorxiv.org/content/10.1101/2023.11.27.568629v1
最近のイメージング解析によって、転写が遺伝子がRNAポリメラーゼIIにより連続して転写される活性状態と、転写が行われない不活性状態との間で切り替わる動的なプロセスであることが明らかになりました。これは転写バーストと呼ばれ、多くの生物種や細胞型で観察される普遍的な現象です。この遷移には、エンハンサーとプロモーター間の相互作用と転写制御因子の集合が関わっていることが示されています。
転写因子は、遺伝子のプロモーター領域や、転写を調節するエンハンサーに結合します。これにより、RNAポリメラーゼIIやBRD4などの転写関連因子が招集されます。これらの因子は多くの天然変性領域(Intrinsically Disordered Regions: IDR)を持ち、凝集体を形成する傾向があります。これが、RNAポリメラーゼによる連続的な転写の開始につながります。しかし、エンハンサーとプロモーター領域の相互作用がどの程度安定しているのかについては、まだ十分には解明されていません。

一方、ライブイメージング、1細胞Hi-C技術、DNA-FISHによる解析が示すところによると、エンハンサーとプロモーターの相互作用を含む高次ゲノム構造は安定したものではなく、むしろ動的に変化することが明らかになっています。例えば、同一の細胞種内でも、個々の細胞における染色体の位置は一定ではなく、細胞によって高次ゲノム構造が動的であり、細胞間の構造の多様性が認識されています。この動的な高次ゲノム構造の動態が転写バーストにどう寄与するは明らかになっていません。

この疑問に答えるために、我々はカリフォルニア工科大学のLong Caiらによって開発されたRNA/DNA/IF-seqFISH技術を採用しました。この技術を用いて、マウスES細胞において、80種類のRNA分子、120のゲノム領域、および20種類のタンパク質及びその翻訳後修飾の局在を同時に可視化しました。
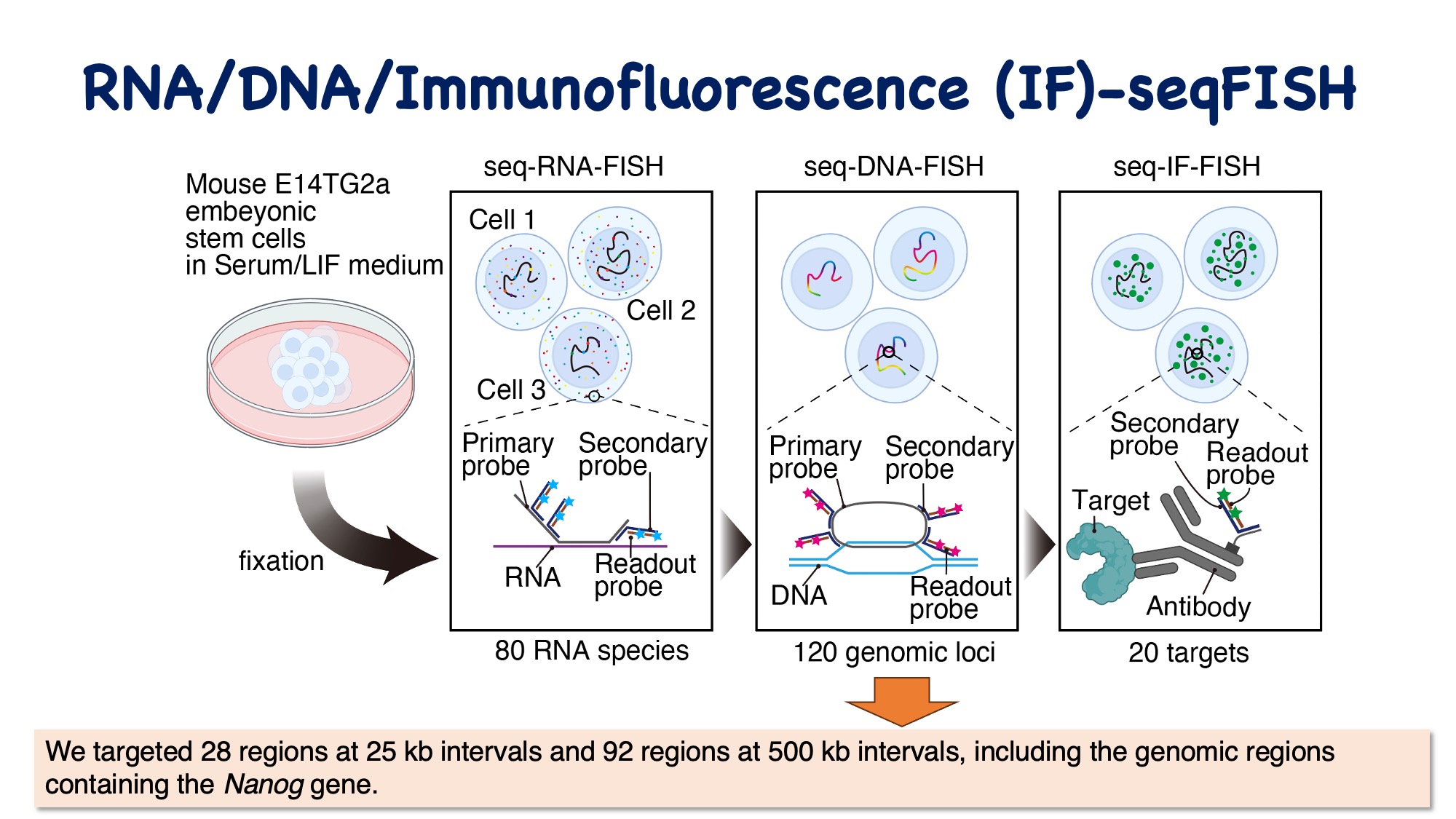
Nanogが活性化状態と不活性化状態にあるときの高次ゲノム構造を比較した結果、構造に差異が存在することが判明しました。特に、転写が活性化された状態では、Nanogの約190 kb上流に位置する遠位エンハンサーがNanog遺伝子の近くに位置していることが明らかになりました。さらに、IF-seqFISHのデータによれば、活性状態の遺伝子周辺には、転写に関わる因子が集約していることが観察されました。これらの因子がエンハンサーとプロモーターの相互作用に影響を与えている可能性が示唆されています。
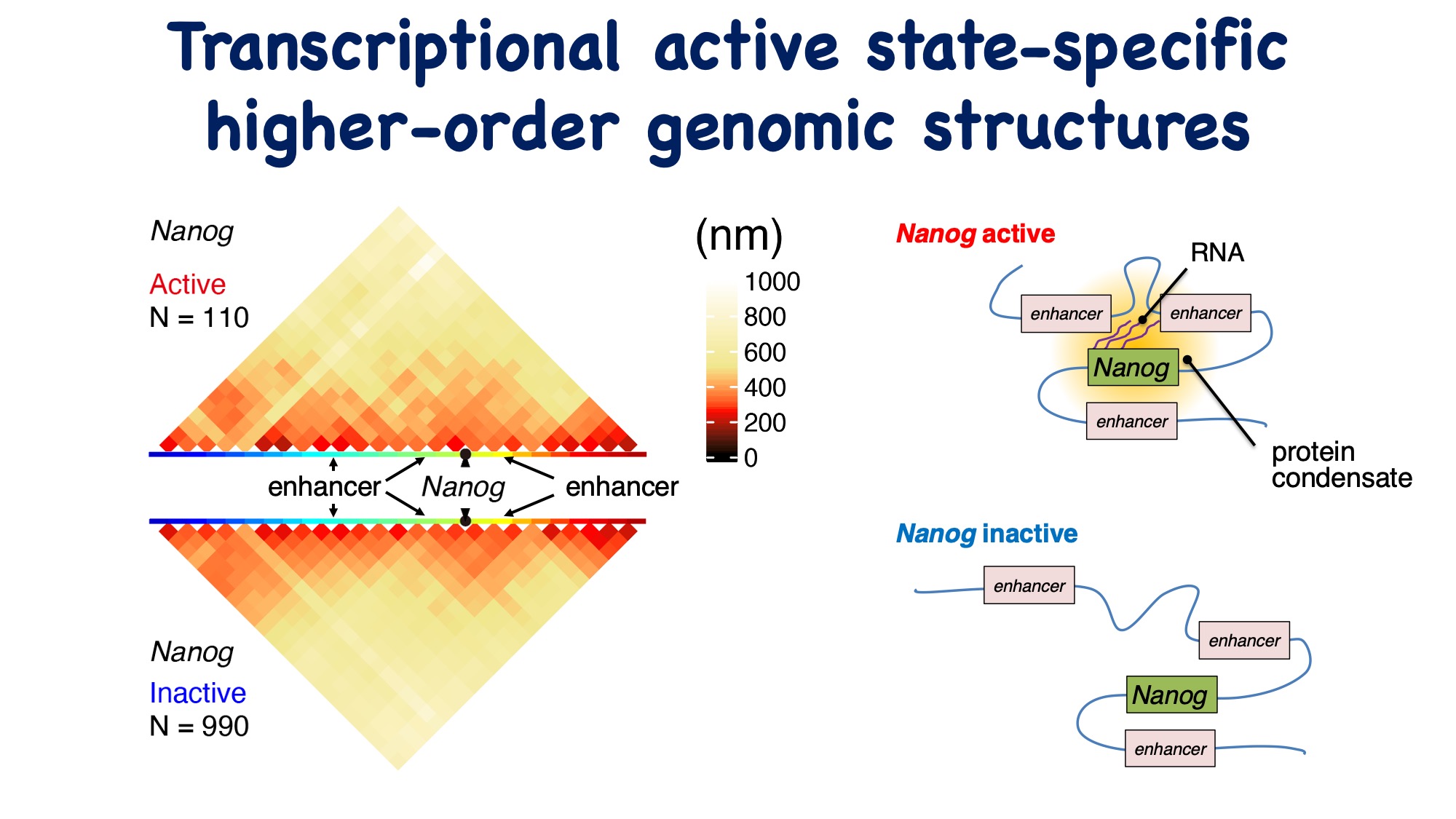
そのため、新海創也さん(理研)が開発したPHi-C技術を活用し、DNA-seqFISHで得られたデータを基に、Nanogのactive/inactive状態における高次ゲノム構造の動態を推定しました。その結果、active状態の際には、inactive状態と比較して遺伝子周辺の粘性が高いことが判明しました。さらに、これによってactive状態において、inactive状態に比べてエンハンサーとプロモーター間の相互作用時間が2倍以上に延長し、エンハンサー・プロモーター間の情報伝達がより効率的に行われていることを示唆しています。